vector nti破解版分享給大家,vector nti advance 11完美破解的喲,包括了詳細的安裝破解教程和crack文件,it貓撲網小編親測可以使用,沒有下載或者不會的趕緊拿走吧。
vector nti advance介紹
Vector NTI是一套功能強大、界面美觀而又友好的分子生物學應用軟件包。它主要包括四個組件,分別對DNA、RNA和蛋白質進行各種分析和操作。

vectorntiadvance11.5.1破解安裝說明
到it貓撲網下載vectorntiadvance11.5.1的安裝程序和破解補丁
先運行安裝程序,這里小編提示,一定要以管理員身份運行,特別是win10、win8等用戶
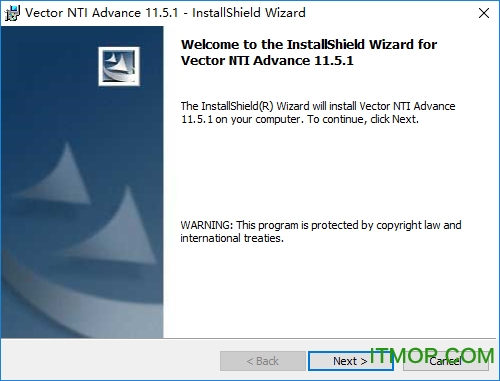
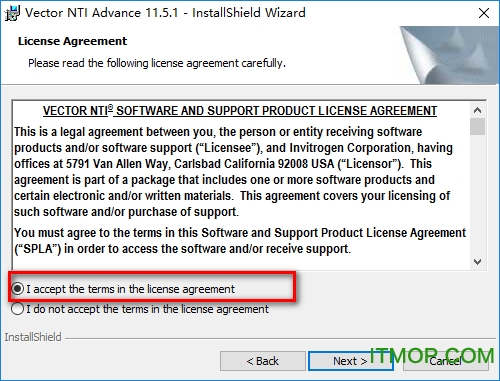
接受許可,繼續安裝
安裝位置大家可以自己選擇,貓撲網只是供測試的,默認在c盤
根據自己的需要選擇vector nti advance安裝類型
稍等一會兒
ok,vector nti advance11.5.1已經安裝完成了,安裝完成后重啟計算機建議
打開選擇的crack文件夾,復制所有的破解文件
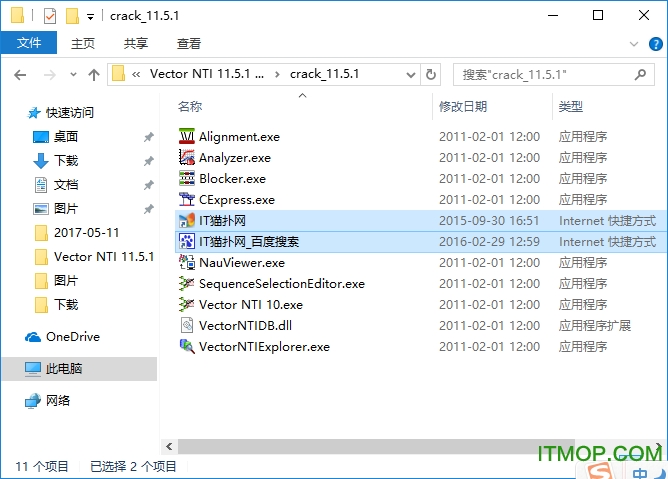
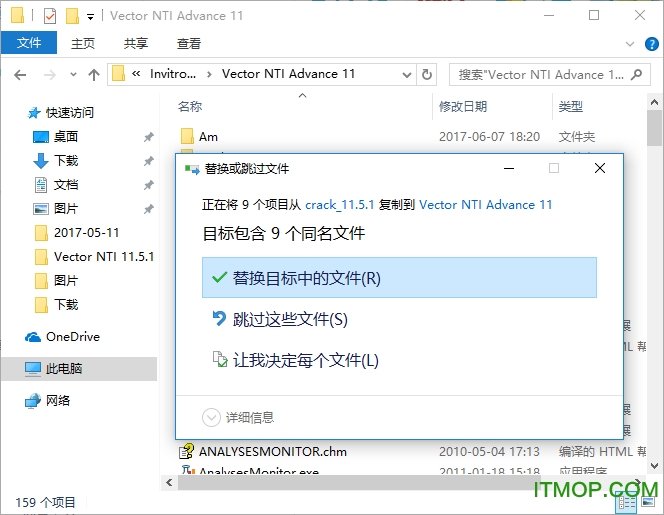
把破解文件復制到安裝后的程序文件夾下(默認為C:\Program Files\Invitrogen\Vector NTI Advance 11\),替換原文件即可。
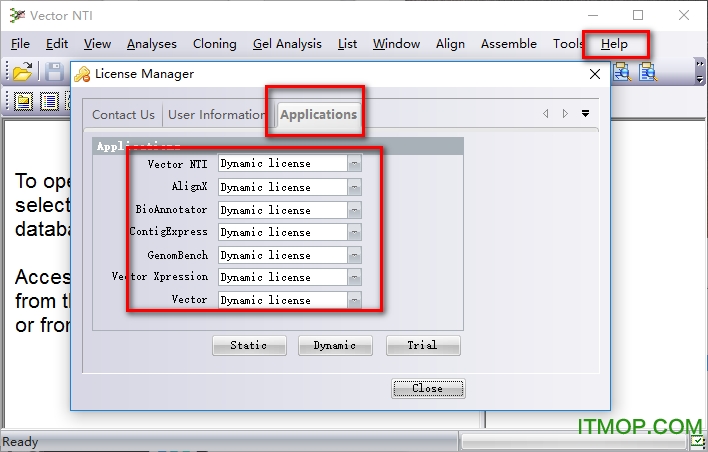
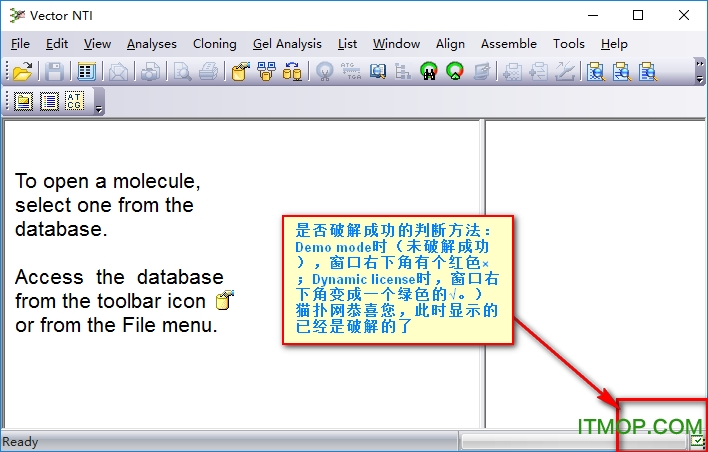
打開vector nti advance,用破解文件替換原文件后,打開Vector NTI,如果發現沒有破解成功,(是否破解成功的判斷方法:Demo mode時(未破解成功),窗口右下角有個紅色×;Dynamic license時,窗口右下角變成一個綠色的√。)請按照以下操作進行
請打開“Help”菜單中的“License Manager”,在Applications選項卡中,將所有程序后面的“Demo mode”,都改為“Dynamic license”。
然后退出軟件,下次打開時就能正常使用了。如下圖右下角已經綠色的勾,說明破解成功了
vector nti advance功能
一,Vector NTI
作為Vector NTI Suite的核心組成部分,它可以在各種分子生物學研究項目的全過程中提供數據組織、編輯和分析支持。
(一)對分子序列的操作
我們以一個DNA序列為例,進行一系列的常規分析;最后將此DNA序列翻譯成氨基酸序列,并對此氨基酸序列進行各種分析。
A,DNA序列為豬生長激素的cDNA序列,長為761bp。首先使用Vector NTI的Create New命令將此序列導入到Vector NTI的數據庫中:
1,第一種方法:如果只知道序列時,點擊Molecule才菜單中的Create New——Using Sequence Editor(DNA/RNA……);
2,在出現的“New DNA/RNA Molecule”對話框中,首先在General填入導入序列的名稱——PGH;
3,在DNA/RNA Molecule活頁中,選中Linear DNA, Animal/other Eukaryotes,Replicon Type中選Chromosome;
4,Description中填入:S.Scrofa Growth hormone mRNA;
5,在Sequence and Maps中點擊“Edit Sequence”按鈕,將DNA序列復制后,點“Paste”按鈕-點“OK”-確認后就可以完成序列導入。
B,如果是一個從GenBank上下載的序列文件,則:點擊 “Molecule” 菜單-Open-Molecule files命令,找到序列文件,在File format中選中GenBank Files;點擊OK。
(二)常規操作:
當序列導入完成后,在桌面出現三個窗口,上左側的窗口中顯示的是該序列的常規信息,上右側窗口則以圖形的格式展示序列的特征區及酶切圖譜等。下面一個窗口顯示的是序列:默認狀態下以雙鏈形式出現,也可以更改為單鏈顯示。
1.選擇序列區域:在圖形區域或序列區域直接拖動鼠標左鍵,同時在最下端的狀態欄中顯示出所選區域的范圍。
2.刪除:選中后直接點擊鍵盤上的Delete鍵,確認后即可刪除。
3.選中序列片段后,點擊Edit菜單,用其中的命令可以完成對此片段的剪切、復制、刪除、定義為新的特征區和用其它序列來代替等。
4.當點在其一特定位置時,我們也可以在此位置插入新的序列:Edit – New – Insert Sequence as
5.當希望序列顯示單鏈時,點擊View – Show Both Strands
(三)常規分析:
1.設計PCR Sequence Primer, Hybridization, Probes:
選中設計引物的模板區或點擊Analyze中的相應命令即可。
需要注意的是,在設計前,首先得將序列存入數據庫中,具體設計由于我們推薦使用Oligo,所以此處不詳述。
2.序列基本信息分析:
選中序列區段后,選Analyze – Oligo Analysis, 在Oligo Analysis對話框中,點擊Analyze按鈕,即可得到分子量、GC含量、Tm值、3‘端的自由能、回文結構及重復序列等基本信息。
3.酶切圖譜分析
點擊Analyze菜單中Restriction Sites命令,出現“Restriction Map Setup”對話框,點擊Add按鈕,填入需要分析的位點,不需要的位點夜可以選中后點Remove按鈕移除。為了顯示正確,我們可以設定超過一定位點數量的酶不顯示,可以限定分析的區域等。點擊OK后程序自動完成酶切分析。
4.Motif查找
點擊Analyze菜單中的Motifs命令,在出現的Motifs Setup對話框中我們可以添加新的Oligo或從Oligo Database和Oligo List中選取;選中后點擊OK按鈕,程序完成Motif的查找,同時給出相似性的百分比。
5.ORF查找
點擊Analyze菜單中的ORF命令,在出現的ORF Setup對話框中,填入ORF的最小長度(多少個密碼子)以及其它一些設定后點擊OK,程序自動完成ORF的查找。
6.翻譯
翻譯前選中一個ORF或一個區域,這里我們希望把pGH cDNA基因完全翻譯成蛋白質,因此選中最長的一個ORF(7-657bp)。點擊Analyze菜單中的Translate命令中的“Into New Protein”-“Direct Strand”,在出現的“New Protein Molecule”對話框中給出新蛋白質的名稱后點擊確定,程序完成翻譯并打開一個新的窗口,顯示氨基酸序列。
7.反翻譯
選中氨基酸序列片斷,點擊Analyze菜單中的BackTranslate命令,確認是“整個序列”還是“僅為選中的序列”后,即可設定簡并度及組織特異性來完成反翻譯。
(三)模擬電泳:
模擬電泳是指對DNA片段進行酶切分析后,通過電腦模擬電泳過程,將酶切片段分離。該功能有利于評估電泳時間,便于驗證實際電泳結果的好壞。
1.點擊Gel菜單 – Create New命令:出現Gel Setup對話框,首先選擇電泳介質的類型-“Agarose Gel”,以及膠的濃度、電壓、膠長和Buffer種類。
2.點擊OK后即打開一個電泳界面,左側是對電泳情況的描述,右側則為膠板。
3.首先配置一個Marker:點Gel菜單 – Create Gel Marker, 出現New Gel Marker對話框, 首先輸入Marker的名稱:pGH-Marker,在Gel Marker活頁中輸入自己設定的片段,100,200,300,400,500,600,1000bp,點擊確定。
4.樣品制備:點Gel菜單 – Create Gel Sample命令出現Create Gel Sample對話框,選擇來源分子SSPGH,選中AvaI和SmaI兩個酶,輸入Sample的名稱SSPGH- AS。此時我們可以把酶切的片段直接加入到膠上,也可以加入到樣品列表中或保存為Marker,我們點擊Add to Gel,則在膠上出現1號樣品。
5.點Marker到膠中:點擊第二行工具欄中的Add Marker Lane圖標,出現Choose Database Gel Marker對話框,選中pGH - Marker,點擊OK,則Marker出現在2道
6.電泳:用鼠標點擊右側窗口激活膠板,點擊第二欄工具欄中的雙箭頭,開始電泳,同時在旁邊顯示電泳時間,再次點擊雙箭頭,電泳停止。
7.計算兩條帶分開時間:選中沒有分開的條帶,點工具欄中的計算器Calculate Seperation Time,即可得到所需信息。
8.導出膠圖:激活膠板,點擊工具欄中的Camera工具,出現Camera對話框,選中需要導出的選項,結果可以到剪貼板,也可以保存成文件。到剪貼板后就可以在Word等文檔編輯器中粘貼。
(四)圖形操作
對于圖形展示的序列信息,我們可以對圖形進行各種修飾和改動,并最終導出需要的圖形,如果序列是質粒,則可得到質粒圖譜。
我們還是以SSPGH序列為例:
1.激活圖形欄,點擊工具欄中的Edit Picture。此時,點擊任意一個需要改動的組件,則鼠標變為四方向箭頭,按住左側則可以任意拖動其位置。
2.點擊左鍵,選中Properties命令,則在Properties對話框中可以改變文字,字體,連線的粗細和顏色。
3.對于特征序列,我們還可改變其填充方式,箭頭方向等。
4.加入注釋:點擊工具欄中的回形針按鈕,出現Annotation對話框,輸入注釋文字(支持中、英文),如:“這是一個測試Sequence”。點擊確定后,文字就出現在圖示窗口中,同樣可以改變其位置、字體、顏色等。
5.修改完成后,點工具欄中的命令,同樣可以到剪貼板或文件。
二.組件AlignX
運行AlignX后出現四個窗口,從上到下,從左到右分別為序列信息窗口,進化樹窗口,同源比較圖示窗口和序列比較窗口。對于同源比較可以分為兩類:一類是兩個或幾個序列(包括DNA/RNA和蛋白質序列)的比較,此時僅限于比較序列的相似性;另一類則是多個序列間的比較并得出系統進化樹。
(一)導入外源序列的方法:點擊Project菜單中Add Files命令,選擇需要比較的序列文件,點擊打開按鈕,確認是DNA/RNA還是Protein Sequence后,點擊Import按鈕就可以完成序列的導入任務。
(二)我們以程序本身提供的演示Project來講述AlignX的使用:
1.選擇Project菜單Open命令,找到Vector NTI的Demo Projects文件夾,打開DNA.apr文件;
2.在文本窗口中,使用鼠標左鍵雙擊任一文件名,就可以得到該文件的所有基本信息;
3.建立一個新的比較策略并進行比較:
①在文本窗口中,按住Ctrl鍵選中四個序列:AF××××
②點擊Alignment菜單中Alignment Setup命令,出現Alignment Setup對話框,在此對話框中有30個選項來確定最終比較結果展示方式,這里我們使用默認值即可。
③點擊Alignment菜單中的Align Selected Sequence命令,片刻后程序完成比較并給出結果和進化樹;
④在View菜單中,我們可以通過Edit Alignment命令來編輯比較結果,使用Display Setup命令來改變結果的展示方式和顏色等。
4.結果的導出:
①進化樹的導出:激活進化樹窗口,點擊工具欄中的Export Tree按鈕即可將進化樹保存為.Ph文件,并可被其他樹編輯軟件所識別。或者點擊Edit菜單中的Camera命令,將圖像保存到剪貼板后在Word等文檔編輯軟件中粘貼;
②序列比較結果的導出;點擊Project菜單中的Export MSF Format命令,將結果保存為.msf文件后,用GeneDoc打開進一步進行編輯和修飾。
三.Contig Express
該組件讓您能夠直接從測序儀或其它文件格式(如GenBank和Fasta等)中導入序列,并將這些阿序列片段拼接成一個長片段。在拼接的過程中可以顯示測序圖譜,可以自動去除載體序列及測序模糊序列。同時能在拼接的完成序列堿基的改動。
1.點擊Project菜單中的Open命令,找到Demo Project文件夾中的DNA.cep文件。當然我們也可以導入自己的序列,方法是點Project菜單中的Add Fragments命令,在此可以導入各種格式的文件。
2.建立拼接策略:點擊Assemble菜單中的Assembly Setup命令,出現Assembly Setup對話框,在此我們使用程序的默認值,不作改動。
3.使用Ctrl鍵將.abi文件全部選中,并點擊Assemble菜單中的Assemble Selected Fragment命令,程序自從完成拼接并出現一個Contig1的圖標。
4.雙擊Contig1圖標,出現另一個窗口展示一個8片段拼接結果。
5.激活序列窗口后,點擊View菜單中的Show All Chromatograms命令,就可以顯示每個序列的測序圖譜。
6.編輯圖譜及序列:如果想改變圖譜或序列上的某個堿基,就可以用框標點擊該堿基,后輸入新的堿基即可。同時,在圖形窗口雙擊任一片段就可以打開一個新的窗口,在此窗口中可以對此序列進行直接的改變了。
7.拼接結果的導出:點擊Edit菜單中的Select All命令選上拼接結果序列,在此點擊Edit菜單,使用Copy命令將拼接結果復制出來。
四.BioPlot
該組件可以完成對DNA、RNA和氨基酸序列的各種理化特性的分析,實際上在DNAStar中有兩個組件來分別完成對DNA/RNA和氨基酸序列的分析,其分析效果并不比BioPlot差,所以在此不再詳述。